생어 염기서열 분석
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
생어 염기서열 분석은 1977년 프레더릭 생어가 개발한 DNA 염기서열 분석 방법으로, 디데옥시뉴클레오타이드(ddNTP)를 사용하여 DNA 복제를 중단시키는 원리를 이용한다. 각 염기에 해당하는 형광 표지된 ddNTP를 사용하여 한 번의 반응으로 네 가지 염기를 모두 판독하는 다이 터미네이터 방식이 널리 사용된다. 생어 염기서열 분석은 비교적 긴 염기서열을 정확하게 읽을 수 있지만, 처리량이 낮고 비용이 많이 든다는 단점이 있다. 이 기술은 질병 진단, 법의학, 농업, 공중 보건 등 다양한 분야에 활용되며, 특히 코로나19 팬데믹 상황에서 바이러스 변이 추적에 중요한 역할을 했다. 생어 염기서열 분석은 프라이머 결합으로 인한 품질 저하, 짧은 DNA 조각만 분석 가능하다는 한계가 있으며, 이를 극복하기 위해 차세대 염기서열 분석(NGS) 기술이 개발되었다.
더 읽어볼만한 페이지
- 분자생물학 기술 - 중합효소 연쇄 반응
DNA 중합 효소를 사용하여 DNA 특정 부위를 복제 및 증폭하는 중합효소 연쇄 반응(PCR)은 유전체 연구, 분자생물학, 법의학, 의학 진단 등 다양한 분야에서 활용되는 기술로, 캐리 멀리스에 의해 고안되어 발전해왔다. - 분자생물학 기술 - 플라스미드
플라스미드는 염색체와 독립적으로 존재하며 자율적으로 증식하는 주로 원형 DNA 형태의 유전 물질로, 유전자 공학에서 유전자 운반체로 사용되며 세균 내에서 항생제 내성 유전자 등을 포함하여 숙주 세포 생존에 기여하고 생명공학 연구 및 유전자 치료에 활용된다. - 분자생물학 - 단백질
단백질은 아미노산 중합체로 생체 구조 유지와 기능에 필수적이며, 아미노산 서열에 따라 고유한 3차원 구조를 형성하여 효소, 구조, 수송, 저장, 수축, 방어, 조절 단백질 등 다양한 기능을 수행하고, 인체 내에서 건강 유지와 질병 예방에 중요한 역할을 하는 필수 영양소이다. - 분자생물학 - 의학
의학은 질병의 진단, 예후, 치료, 예방을 연구하는 과학 및 실천 분야이며, 고대부터 발전하여 현대에는 다양한 전문 분야로 세분화되고 첨단 기술 발전에 따라 혁신적인 변화를 겪고 있다.
2. 방법
고전적인 사슬 종료 방법은 DNA 염기서열 분석을 위해 단일 가닥 DNA 주형, DNA 프라이머, DNA 중합 효소, 정상적인 dNTPs, 그리고 변형된 ddNTPs를 필요로 한다. ddNTP는 DNA 가닥 연장을 종결시키는데, 이는 3'-OH기가 없어 DNA 중합 효소가 DNA 연장을 중단시키기 때문이다. ddNTP는 자동 염기 서열 분석 장치에서 검출될 수 있도록 방사성 또는 형광으로 표지된다.[23]
DNA 샘플은 네 개의 개별 염기 서열 분석 반응으로 나뉘며, 각 반응에는 네 가지 디데옥시리보뉴클레오티드 (ddATP, ddGTP, ddCTP, ddTTP) 중 하나만 추가된다. ddNTP는 상응하는 dNTP보다 약 100배 적은 농도로 추가되어 전체 염기서열이 계속 복제되는 동안 충분한 조각이 만들어지도록 한다. 이후, 생성된 DNA 단편은 열 변성되고 젤 전기 영동을 사용하여 크기별로 분리된다.
1977년 발표된 원본 방식에서는 ssDNA의 염기쌍 고리 형성이 밴드 분리에 어려움을 야기했다. 이는 변성 폴리아크릴아미드-요소 젤을 사용하여 네 개의 개별 레인(A, T, G, C)에서 각 반응을 실행하여 해결했다. DNA 밴드는 자동 방사선 촬영 또는 UV 광선으로 시각화되며, DNA 염기 서열은 X선 필름 또는 젤 이미지에서 직접 판독할 수 있다.
사슬 종결 염기 서열 분석의 기술적 변형에는 방사성 동위 원소 표지를 위한 방사성 인을 포함하는 뉴클레오티드로 표지하거나, 5' 말단에 형광 염료로 표지된 프라이머를 사용하는 것이 있다. 염료-프라이머 염기 서열 분석은 빠르고 경제적인 분석 및 자동화를 위해 광학 시스템에서 읽기를 용이하게 한다. 리로이 후드와 동료들은 형광 표지된 ddNTP 및 프라이머를 개발하여 자동화된 고처리량 DNA 염기 서열 분석의 기반을 마련했다.[7][8]
사슬 종결 방법은 DNA 염기 서열 분석을 크게 단순화했으며, 현재 상업적으로 이용 가능한 키트도 판매되고 있다. 그러나 프라이머의 비특이적 결합이나 DNA 2차 구조는 염기 서열의 정확도에 영향을 줄 수 있다.
2. 1. 생어 염기서열 분석 (Sanger Sequencing)
생어 염기서열 분석(Sanger Sequencing)은 1977년 프레더릭 생어(Frederick Sanger)가 개발한 DNA 염기서열 분석 방법이다. 이 방법은 디데옥시뉴클레오타이드(ddNTP)를 이용하여 DNA 복제를 중단시키는 원리를 이용한다. ddNTP는 DNA 중합효소가 DNA 가닥을 연장하는 데 필요한 3'-OH기가 없어, 특정 염기에서 DNA 복제를 멈추게 한다.[23]DNA 샘플은 네 가지 반응으로 나뉘는데, 각 반응에는 다른 ddNTP(ddATP, ddGTP, ddCTP, ddTTP)가 첨가된다. 각 반응에서 생성된 다양한 길이의 DNA 단편들은 겔 전기영동(gel electrophoresis) 또는 모세관 전기영동(capillary electrophoresis)을 통해 크기별로 분리된다. 분리된 DNA 단편들의 형광 신호를 검출하여 원래 DNA의 염기서열을 판독한다.
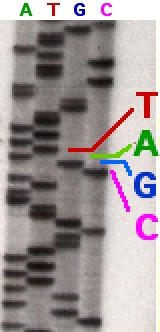
오른쪽 사진은 방사성 표지된 시퀀싱 겔의 일부를 보여준다. 어두운 띠는 다양한 길이의 DNA 조각에 해당하며, 한 레인의 어두운 띠는 특정 ddNTP 삽입으로 인한 사슬 종료의 결과이다. 네 레인의 띠 위치를 비교하여 DNA 염기서열을 읽을 수 있다.
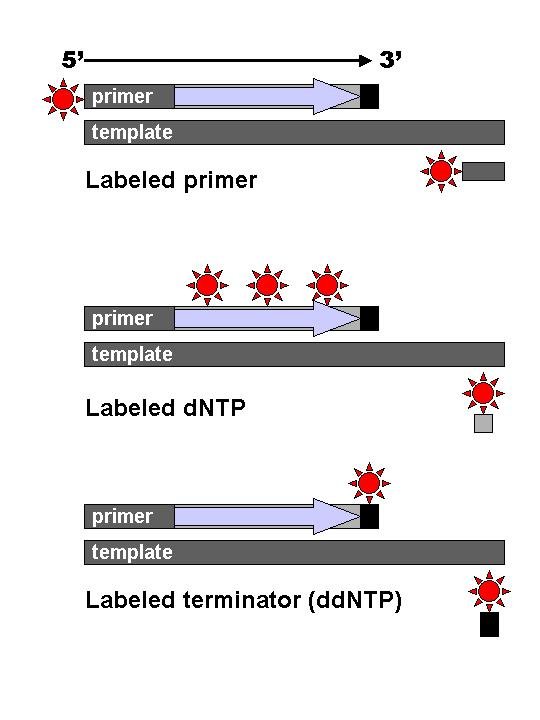
초기에는 방사성 동위원소 표지를 사용했지만, 리로이 후드와 동료들이 형광 표지된 ddNTP 및 프라이머를 개발하면서 자동화된 고처리량 DNA 염기서열 분석이 가능해졌다.[7][8]
생어 염기서열 분석은 비교적 긴 염기서열(약 500-900bp)을 정확하게 읽을 수 있다는 장점이 있지만, 한 번에 처리할 수 있는 양이 적고 비용이 많이 든다는 단점이 있다.
2. 1. 1. 자동화 및 샘플 준비
자동화된 DNA 염기서열 분석 장비(DNA 시퀀서)는 모세관 전기 영동을 사용하여 크기(또는 길이)별로 DNA 가닥을 분리하고, 염료 형광을 감지 및 기록하며, 형광 피크 추적 크로마토그램으로 데이터를 출력한다.[9] 시퀀싱 반응(열 순환 및 표지), 샘플의 정제 및 완충 용액 재현탁은 시퀀서에 샘플을 넣기 전에 별도로 수행한다.[9] 이러한 DNA 시퀀서는 한 번에 최대 384개의 DNA 샘플을 분석할 수 있으며, 하루에 최대 24번 실행할 수 있다.[9]다이 터미네이터 염기서열 분석(Dye-terminator sequencing)은 사슬 종결자 ddNTP에 형광 표지를 부착하여, 네 번의 반응이 아닌 한 번의 반응으로 염기서열 분석을 가능하게 하는 방법이다.[9] 이 방법에서는 네 가지 디데옥시뉴클레오티드 사슬 종결자 각각에 서로 다른 파장의 빛을 방출하는 형광 염료가 표지되어 있다.[9] 더욱 편리하고 빠르기 때문에, 다이 터미네이터 염기서열 분석은 현재 자동 염기서열 분석의 주류를 이루고 있다.[9]
2. 1. 2. 한계
생어 염기서열 분석은 처음 15-40개 염기 부분에서 프라이머 결합 문제로 인해 품질이 저하될 수 있다. 또한 700-900개 염기 이후에도 염기서열 분석 결과의 품질이 떨어지는 경향이 있다.[12][13] Phred와 같은 염기서열 판독 소프트웨어는 염기서열의 품질을 추정하여 품질이 낮은 부분을 제거하는 데 도움을 준다.[12][13]DNA 2차 구조도 염기서열 정확도에 영향을 미치는 요인이다. DNA 조각을 염기서열 분석하기 전에 클로닝을 하는 경우에는 생성된 염기서열에 클로닝 벡터의 일부가 포함될 수 있다는 한계도 있다.
현재 생어 염기서열 분석 방법으로는 한 번의 반응으로 300-1000개 뉴클레오티드 정도의 짧은 DNA 조각만 직접 염기서열 분석할 수 있다. 이보다 긴 DNA 조각 분석이 어려운 주요 원인은 단 하나의 뉴클레오티드 길이 차이만 나는 큰 DNA 조각들을 분리해낼 수 있는 충분한 분리 능력이 부족하기 때문이다.
2. 2. 미세유체 생어 염기서열 분석 (Microfluidic Sanger Sequencing)
미세유체 생어 염기서열 분석은 DNA 염기서열 분석을 위한 랩온어칩 응용 분야로, 생어 염기서열 분석 단계(열 순환, 시료 정제, 모세관 전기 영동)를 나노리터 규모의 시료 부피를 사용하여 웨이퍼 크기 칩에 통합한 기술이다.[16] 이 기술은 긴 길이의 정확한 서열을 생성하며, 생어 염기서열 분석 단계를 통합하고 자동화함으로써 기존 생어 방식의 단점(예: 고가의 시약 과다 소비, 고가 장비 의존, 인력 집약적인 조작 등)을 없앤다.현대적인 고처리량 유전자 염기서열 분석은 유전체를 작은 단일 가닥 조각으로 분쇄한 다음, 중합효소 연쇄 반응(PCR)을 통해 조각들을 증폭하는 것을 포함한다. 생어 방식을 채택하면, 각 DNA 조각은 형광 표지된 디데옥시 사슬 종결 뉴클레오타이드를 통합하여 영구적으로 종결되므로, 각 조각의 길이가 1개의 염기만큼 다르고 말단 염기에 염기 특이적 형광 표지가 있는 DNA "래더"가 생성된다. 증폭된 염기 래더는 자동화된 ''in situ'' 형광 표지 ssDNA 조각의 "결승선" 검출을 통해 모세관 어레이 전기 영동(CAE)으로 분리되며, 이는 조각의 정렬된 염기서열을 제공한다. 이 염기서열 판독은 컴퓨터로 조립되어 중복되거나 인접한 염기서열("컨티그"라고 함)로 구성되며, 완전히 조립되면 전체 유전체 염기서열과 유사해진다.[16]
생어 방식은 최대 약 800 bp(일반적으로 비농축 DNA의 경우 500–600 bp)의 판독 길이를 얻는다. 생어 방식의 더 긴 판독 길이는 특히 유전체의 반복 영역을 염기서열 분석하는 측면에서 다른 염기서열 분석 방식에 비해 상당한 이점을 보인다. 짧은 판독 염기서열 데이터의 문제는 특히 새로운 유전체 염기서열 분석 ''(de novo)'' 및 고도로 재배열된 유전체 세그먼트, 일반적으로 암 유전체 또는 구조적 변이를 나타내는 염색체 영역에서 나타난다.[18]
아폴로 100 플랫폼(Microchip Biotechnologies Inc., 더블린, 캘리포니아)([17])은 생어 염기서열 분석의 처음 두 단계(열 사이클링 및 정제)를 완전 자동화된 시스템에 통합한다. 제조업체는 시료와 시약이 시스템에 로드된 후 3시간 이내에 모세관 전기 영동을 수행할 수 있다고 주장한다. 아폴로 100 플랫폼은 마이크로리터 미만의 시약 부피를 필요로 한다.
2. 3. 차세대 염기서열 분석 (Next-Generation Sequencing, NGS)
차세대 염기서열 분석(Next-Generation Sequencing, NGS)은 생어 염기서열 분석의 한계를 극복하기 위해 개발된 대용량 병렬 염기서열 분석 기술이다. 수많은 DNA 단편들을 동시에 병렬적으로 처리하여, 짧은 시간에 대량의 염기서열 데이터를 생산할 수 있다. NGS 기술은 유전체 전체 염기서열 분석(whole genome sequencing), 전사체 분석(transcriptome analysis), 후성유전체 분석(epigenome analysis) 등 다양한 분야에 활용되고 있다.[16]생어 방식과 차세대 방식을 포함한 유전자 서열 분석 기술의 성능은 다음과 같다.[18][19][20]
DNA 염기서열 분석 기술은 기초 생명과학 연구뿐만 아니라, 다양한 응용 분야에서 활용되고 있다.
고처리량 시퀀싱의 궁극적인 목표는 저렴하고 확장된(더 긴) 읽기 길이를 얻는 데 매우 효율적인 시스템을 개발하는 것이다. 각 단일 전기 영동 분리의 더 긴 읽기 길이는 de novo DNA 시퀀싱과 주어진 중복성에서 DNA 컨티그를 시퀀싱하는 데 필요한 템플릿 수를 실질적으로 줄여준다. 미세 유체 공학은 더 빠르고, 저렴하며, 쉬운 시퀀스 조립을 가능하게 할 수 있다.[16]
3. 응용
4. 한계
Sanger sequencing영어은 약 300~1000 염기 정도의 비교적 짧은 DNA 조각만 처리할 수 있다는 한계가 있다. 첫 15~40 염기 부분과 700~900 염기 이후 부분에서는 판독 품질이 저하될 수 있다.[1]
참조
[1]
논문
Battle of the DNA sequencers
1988
[2]
논문
A multiple-capillary electrophoresis system for small-scale DNA sequencing and analysis
1999-12-15
[3]
논문
Next-generation DNA sequencing
2008-10
[4]
논문
A Sanger sequencing protocol for SARS-CoV-2 S-gene
2021-11
[5]
논문
Novel surveillance network for norovirus gastroenteritis outbreaks, United States
2011-08
[6]
논문
DNA sequencing with chain-terminating inhibitors
1977-12
[7]
논문
Fluorescence detection in automated DNA sequence analysis
[8]
논문
The synthesis of oligonucleotides containing an aliphatic amino group at the 5' terminus: synthesis of fluorescent DNA primers for use in DNA sequence analysis
1985-04
[9]
논문
Guidelines for Sanger sequencing and molecular assay monitoring
2020-11
[10]
뉴스
A Timeline of the Coronavirus Pandemic
https://www.nytimes.[...]
2021-12-05
[11]
논문
Recent advances in SARS-CoV-2 Spike protein and RBD mutations comparison between new variants Alpha (B.1.1.7, United Kingdom), Beta (B.1.351, South Africa), Gamma (P.1, Brazil) and Delta (B.1.617.2, India)
2021-09
[12]
웹사이트
Phred – Quality Base Calling
http://www.phrap.com[...]
2011-02-24
[13]
논문
Base-calling for next-generation sequencing platforms
2011-09
[14]
논문
Sequencing of genomic DNA by combined amplification and cycle sequencing reaction
2005-01
[15]
논문
SeqSharp: A general approach for improving cycle-sequencing that facilitates a robust one-step combined amplification and sequencing method
2010-05
[16]
논문
DNA sequencing and genotyping in miniaturized electrophoresis systems
2004-11
[17]
간행물
Apollo 100
http://www.microchip[...]
Microchip Biologies Inc.
[18]
논문
Applications of next-generation sequencing technologies in functional genomics
2008-11
[19]
논문
High resolution DNA separations using microchip electrophoresis
2007-07
[20]
논문
Next-Generation Sequencing and Emerging Technologies
2019-10
[21]
논문
Applications of long-read sequencing to Mendelian genetics
2023-06
[22]
논문
MinION-based long-read sequencing and assembly extends the ''Caenorhabditis elegans'' reference genome
2018-02
[23]
논문
DNA sequencing with chain-terminating inhibitors
1977-12
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com

